论坛简介:
计算生物信息学研究所是西部第一个致力于研究计算机科学、数学学科与生命学科的交叉学科的研究所。研究方向包括:机器学习与海量数据挖掘基础理论与方法;高通量组学数据挖掘及在复杂疾病中的应用;生物医学数据建模与分析高效算法,数据分析平台及可视化软件。核心任务是围绕医疗健康大数据(组学数据,影像数据,临床数据等),开展人工智能、统计计算、数据挖掘、模式发现等理论和关键技术研究,以揭示数据蕴含的生物及医学意义,并建立智能辅助诊疗、药物发现等方法与平台。
为了紧跟学术发展前沿,加强计算机领域院友教师间的学术交流,在我校90周年校庆之际,特邀请西北工业大学、曲阜师范大学、中兴通讯无线研究院、西安交通大学、西安理工大学、山东大学、山东工商学院等高校和企业的优秀院友,通过主题报告的形式,分享计算机领域理论和技术的新进展,以加强学术前沿交流,促进我校计算机学科更好地发展,扩大学科影响力与知名度。论坛由西安电子科技大学计算生物信息学研究所主办,以线下+线上的形式开展。
论坛组织
主办:
西安市计算生物信息学研究所重点实验室
承办:
计算生物信息学研究所
论坛主席:
高琳 教授
论坛副主席:
袁细国 副教授
论坛安排
论坛时间:2021年9月19日
论坛形式:线下+线上
线上参与方式:腾讯会议会议号:812 482 530
论坛地点:北校区主楼2区319
议程安排
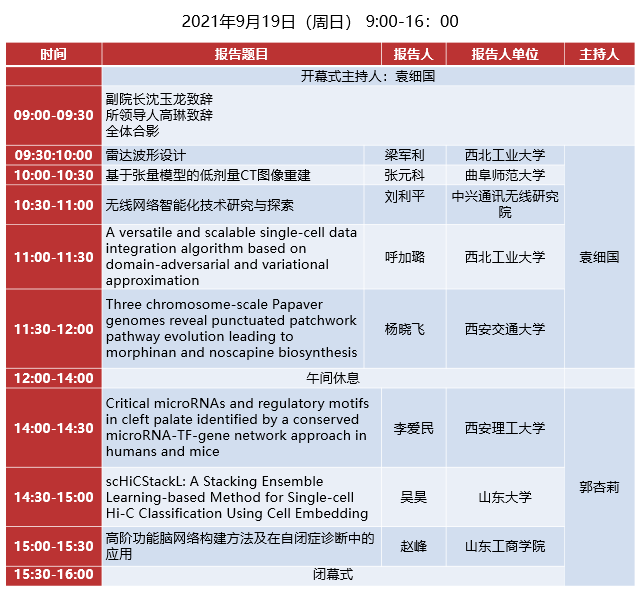
报告嘉宾简介
梁军利
西北工业大学
太阳集团www0638院友

报告题目:雷达波形设计
报告摘要:
相比较以往的接收端信号处理技术,波形设计是从雷达发射端便开始积极争取赢得主动的信号处理技术。随着高速DAC器件、任意波形产生器的应用,以及现代优化计算手段的提高,使得设计和发射任意复杂波形成为可能。本报告的内容为:结合波形的恒模、频谱赋形、相关特性、模糊函数、发射方向图等特性,设计复杂探测波形以适应复杂电磁场环境感知的需要。
报告人简介:
梁军利,博士,教授,博士生导师。2004年在西安电子科技大学获得硕士学位,2007年在中国科学院大学获得博士学位,2012-2013年在美国佛罗里达大学做博士后。2014年和2019年在香港城市大学进行高级学术访问。现在西北工业大学电子信息学院工作。主要研究方向为:雷达信号处理,6G,雷达通信一体化,机器学习与图像处理,非凸优化及其应用等。作为第1或通信作者在IEEE TSP,TAP,TIP,TAP,TNNLS,TAES,ELSEVIER:SP期刊发表学术论文40余篇。
张元科
曲阜师范大学
太阳集团www0638院友

报告题目:基于张量模型的低剂量CT图像重建
报告摘要:
计算机断层成像(CT)是临床重要诊断方式,但其产生的辐射极大增加人体患病风险。低剂量CT技术能有效减少辐射,但其成像质量会严重下降,对低剂量CT的有效成像具有重要意义。基于张量模型的低剂量CT图像重建研究将张量模型引入低剂量CT重建领域,通过对CT影像进行张量建模并用于目标成像,以达到成像质量提升目的。本报告主要汇报了近期两项主要工作,包括:(1)建立了一种新的张量低秩变换子空间稀疏约束模型以描述脑灌注影像序列帧间造影剂浓度结构各向异性特性。基于该模型,提出了一种低剂量脑灌注CT图像重建算法,实现了低剂量脑灌注CT的高质量成像。(2)提出了一种新的加权张量低秩和可学习分析稀疏表示混合约束模型,以更精确表征CT图像中非局部相关性质和局部变换稀疏性质。基于该模型,提出了一种新的低剂量扇形束CT重建算法,实验表明所提算法在噪声抑制和纹理特征保持上优于当前同类算法。
报告人简介:
张元科,1979年出生,山东蓬莱人,博士,副教授。2011年6月于西安电子科技大学计算机科学学院获工学博士学位,2016-2020年分别进入空军军医大学(原第四军医大学)生物医学工程博士后流动站、南方医科大学临床医学博士后流动站从事博士后研究工作。主要从事医学图像成像与分析的相关研究工作,主持完成国家自然科学基金、中国博士后科学基金项目、山东省自然科学基金等多项;先后在国内外重要学术刊物与国际会议上发表论文50余篇,其中以第1作者在包括《IEEE Transactions on Medical Imaging》(SCI,IF=10.048)、《IEEE Transactions on Computational Imaging》(SCI,IF= 3.49)、《IEEE Transactions on Nuclear Science》(SCI,IF= 1.679)、《Medical Physics》(SCI,2区,4.071)、《电子学报》(EI)等医学图像成像与分析领域顶级/权威期刊发表论文多篇。《IEEE Transactions on Medical Imaging》、《Medical Physics》、《IEEE Access》等多个国内外期刊审稿人。
刘利平
中兴通讯无线研究院副院长
太阳集团www0638院友

报告题目:无线网络智能化技术研究与探索
报告摘要:
无线通信网络加持AI,基于大数据分析和深度学习,实现高复杂性多维分析,有限时间内寻求到最优解,使得通信网络开启智能时代。5G时代建网智能规划、智能运维、AI赋能天线权值自优化、智能网络编排、智能用户编排、智能节电、智慧运营等。本报告对未来无线网络智能化相关网络架构、研究方向、关键技术等展开探讨探索。
报告人:
刘利平,2004年硕士毕业于西安电子科技大学计算机学院,同年加入中兴通讯西安研发中心,历任研发工程师、研发部长、日本分公司CTO、无线研究院副院长,主管无线产品预研、算法研究及相关领域4G/5G产品开发,同步负责欧洲、中国大陆、亚洲等重点市场4G/5G项目技术交付。
呼加璐
西北工业大学
太阳集团www0638院友

报告题目:A versatile and scalable single-cell data integration algorithm based on domain-adversarial and variational approximation
报告摘要:
Single-cell technologies provide us new ways to profile transcriptomic landscape, chromatin accessibility, spatial expression patterns in heterogeneous tissues at the resolution of single cell. With enormous generated single-cell datasets, a key analytic challenge is to integrate these datasets to gain biological insights into cellular compositions. Here, we developed a domain-adversarial and variational approximation, DAVAE, which can integrate multiple single-cell datasets across samples, technologies and modalities with a single strategy. Besides, DAVAE can also integrate paired data of ATAC profile and transcriptome profile that are simultaneously measured from a same cell.With a mini-batch stochastic gradient descent strategy, it is scalable for large-scale data, and can be accelerated by GPUs. Results on seven real data integration applications demonstrated the effectiveness and scalability of DAVAE in batch-effect removing, transfer learning, and cell type predictions for multiple single-cell datasets across samples, technologies and modalities.DAVAE was implemented in a toolkit package scbean in the pypi repository, and the source code can be also freely accessible athttps://github.com/jhu99/scbean.
报告人简介:
呼加璐,博士,西安电子科技大学本科硕士毕业(导师:高琳教授),德国柏林自由大学生物信息学博士,美国密歇根大学博士后,现任西北工业大学计算机学院副教授博导。一直致力于多组学数据的分析工作,主持国家自然基金项目两项,省部级项目三项,发表SCI文章十多篇,其中代表作曾在Bioinformatics,Briefings in Bioinformatics, BMC Medicines等国际知名期刊上发表。
杨晓飞
西安交通大学
太阳集团www0638院友

报告题目:Three chromosome-scale Papaver genomes reveal punctuated patchwork pathway evolution leading to morphinan and noscapine biosynthesis
报告摘要:
For millions of years, plants have evolved a plethora of structurally diverse secondary metabolites (SM) to support their sessile lifestyles through continuous biochemical pathway innovation. While new genes commonly act as drivers of SM pathway evolution in plants, little is known about the evolution of a full biosynthetic pathway. Models for pathway evolution involve recruitments of new genes forwardly, backwardly, or in a patchwork manner along the cascade reactions. With three chromosome-scale Papaver genome assemblies, we revealed different numbers of whole genome duplications (WGDs) (zero in P. rhoeas, one in P. somniferum and two in P. setigerum), which accelerated post-WGD chromosomal rearrangements with a nonrandom distribution towards SM optimization. Furthermore, a burst of structural variants involving fusion, translocation and duplication during the past 7.7 million years assembled nine new genes from loci of low and non-tissue-specific expression to a locus stem-specifically expressed to form the benzylisoquinoline alkaloid gene cluster, following a punctuated patchwork model. Finally, we showed that total expression of all gene copies besides copy numbers of biosynthetic genes contributed to morphinan production. Our results demonstrate how a set of new genes were recruited from a WGD-induced repertoire of unregulated enzymes with promiscuous reactivities to form a novel pathway with spatiotemporal constraint like a metabolon.
报告人简介:
杨晓飞,博士,现为西安交通大学太阳集团www0638副教授,博士生导师,计算机学会生物信息专委会委员。博士毕业于西安电子科技大学计算机应用技术专业,研究方向为数据挖掘,生物信息学,博士毕业论文荣获2019年陕西省优秀博士论文,2019年西安电子科技大学优秀博士论文。于2018年10月至2019年10月以国家公派访问学者的身份赴美国杰克逊实验室进行为期一年的访问学习。主要从事多组学数据分析及算法开发,包括基因组组装及注释、结构变异检测算法开发、表观基因组致病模式挖掘、癌症基因组数据分析等研究。主持国家自然科学基金面上项目、青年科学基金项目、中央高校基本科研业务费、中国博士后面上项目、陕西省博士后项目、国家科技重大专项(子课题负责人)等项目,累计主持科研经费130余万元。共发表学术论文20余篇(第一或通讯作者共10篇),包括Science, Genome Biology, Briefings in Bioinformatics, Bioinformatics,BMC Genomics,Genomics Proteomics & Bioinformatics,Quantative Biology 等期刊,所发表论文累计引用759余次,最高单篇引用171次(Google Scholar),h-index为12,i10指数为12.
李爱民
西安理工大学
太阳集团www0638院友

报告题目:Critical microRNAs and regulatory motifs in cleft palate identified by a conserved microRNA-TF-gene network approach in humans and mice
报告摘要:
Cleft palate (CP) is the second common congenital birth defect. The etiology of CP is complicated with the involvement of various genetic and environmental factors. To identify the gene regulatory mechanisms, we designed a powerful regulatory analytical approach to find the conserved regulatory networks between humans and mice, from which we identified critical microRNAs (miRNAs), target genes, and regulatory motifs related to CP. We constructed miRNA-transcription factor (TF)-gene networks in both humans and mice. We found a consensus regulatory loop (miR17/miR20a-FOXE1-PDGFRA) and pinpointed eight microRNAs in both humans and mice. The role of miR-140, which had the strongest association with CP, was investigated in both human and mouse palate cells. We found that miR-140-5p overexpressionsignificantly inhibited cell proliferation in these cells. Our results indicated that miR-140-5p overexpression suppressed the expression of BMP2 and FGF9 in cultured human palate cells and Fgf9 and Pdgfra in cultured mouse palate cells.
报告人简介:
李爱民,西安理工大学计算机科学与工程学院讲师,西安电子科技大学计算机学院博士。中国计算机学会会员,国家留学基金委公派访问学者。主要研究方向为计算生物信息学、机器学习。发表学术论文30余篇,其中SCI论文22篇;以第一作者或共同第一作者发表SCI论文11篇,其中2篇1区Top期刊论文,1篇ESI 1%高被引论文,最高影响因子11.147。担任多个著名生信期刊和会议的编辑和审稿人。获发明专利1项,受理专利6项。主持或参与科研项目20余项,其中主持省部级项目2项。指导学生获得国家级大创1项。
吴昊
山东大学
太阳集团www0638院友

报告题目:scHiCStackL: A Stacking Ensemble Learning-based Method for Single-cell Hi-C Classification Using Cell Embedding
报告摘要:
Single-cell Hi-C data is a common data source for studying the differences in the three-dimensional structure of cell chromosomes. The development of single-cell Hi-C technology makes it possible to obtain batches of single-cell Hi-C data. How to quickly and effectively discriminate cell types has become one hot research field. However, the existing computational methods to predict cell types based on Hi-C data are found to be low in accuracy. Therefore, we propose a high accuracy cell classification algorithm, called scHiCStackL, based on single-cell Hi-C data. In our work, we first improve the existing data preprocessing method for single-cell Hi-C data, which allows the generated cell embedding better to represent cells. Then, we construct a two-layer stacking ensemble model for classifying cells. Experimental results show that the cell embedding generated by our data preprocessing method increases by 0.23%, 1.22%, 1.46% and 1.61% comparing to the cell embedding generated by the previously published method scHiCluster, in terms of the Acc, MCC, F1 and Precision confidence intervals, respectively, on the task of classifying human cells in the ML1 and ML3 datasets. When using the two-layer stacking ensemble framework with the cell embedding, scHiCStackL improves by 13.33%, 19%, 19.27% and 14.5% over the scHiCluster, in terms of the Acc, ARI, NMI and F1 confidence intervals,respectively. In summary, scHiCStackL achieves superior performance in predicting cell types using the single-cell Hi-C data. The webserver and source code of scHiCStackL is freely available at/index.htmland https://github.com/HaoWuLab-Bioinformatics/scHiCStackL, respectively.
报告人简介:
吴昊,博士,硕士生导师,山东大学软件学院副教授;CCF高级会员、BIIP专委委员、NCCBB专委委员、陕西生物信息学学会理事;曾获得校级青年教师讲课比赛二等奖、校级优秀教师、优秀指导教师、校级教学成果二等奖等荣誉。近五年主持国家自然科学基金面上项目、省部级、局厅级等项目10余项。近年来,在《Briefings in Bioinformatics》、《Engineering Applications of Artificial Intelligence》、《计算机学报》等国内外期刊上发表学术论文30余篇;申请国家发明专利4项,软件著作权9项,出版教材和专著3本。研究方向:生物大数据挖掘、人工智能、计算生物信息学、生命健康等。
赵峰
山东工商学院
太阳集团www0638院友

报告题目:高阶功能脑网络构建方法及在自闭症诊断中的应用
报告摘要:
自闭症(Autism spectrum disorder:ASD,自闭症谱系障碍)是一种发病率较高的神经发育障碍性儿童疾病,无法根治,有效的治疗方法也不普遍,使得许多患者的家庭无处求援。诸多研究表明ASD可引起神经形态学方面的改变,意味着ASD儿童与正常儿童(Normal)相比,其神经影像上存在差异。因此,如何挖掘、利用神经影像上的差异,辅助医生进行临床诊断成为当前智慧医疗领域的研究热点,对儿童患者走上比较正面的生活轨道意义重大。本报告重点介绍如何基于静息态磁共振数据,构建用于反映各个功能脑区之间深层次连接关系的高阶网络的方法,为深入理解脑神经活动规律、探索包括ASD在内容的诸多脑疾病的病因和辅助诊断提供技术支持。
报告人简介:
赵峰,山东工商学院太阳集团www0638教授,副院长,硕士研究生导师,中国图学学会大数据专委会委员,山东工商学院“凤凰拔尖人才”、“科研标兵”,清华大学自动化系访问学者,美国北卡罗来纳大学教堂山分校医学影像中心访问学者。主要研究方向包括:人工智能、机器学习、医学图像分析、金融大数据分析。曾获山东高等学校优秀科研成果奖二等奖、三等奖各1项;在《Expert Systems with Applications》、《Brain Imaging and Behavior》等国内外学术期刊上发表论文20余篇;出版学术专著1部;主持国家自然科学基金2项,省自然科学基金2项。
高琳 论坛主席 西安电子科技大学

个人简介:高琳,女,博士,二级教授,博士生导师,太阳集团www0638。在计算生物信息学、数据挖掘与机器学习、图论与组合优化算法方面进行了长期研究,承担国家自然科学基金重点项目3项、重大研究计划等项目,参与了科技部精准医疗重点专项。研究成果获陕西省自然科学技术一等奖,电子学会自然科学技术二等奖,单细胞环状伪轨迹推断算法入选“2019年中国生物信息学十大算法与工具”,在Nature Communications,Science Advances,Advanced Science,Nucleic Acids Research, Bioinformatics ,Briefings in Bioinformatics等期刊发表论文100余篇。
创办了计算生物信息学研究所,建立了西安市计算生物信息学重点实验。中国人工智能学会理事,中国计算机学会“生物信息专委员会”副主任,中国人工智能学会“生物信息学与人工生命专业委员会”副主任,中国运筹学会“计算生物信息学分会”副理事长。陕西省学位委员会学科评议组第三届成员。西安电子科技大学第八届学术委员会委员。
袁细国 论坛副主席 太阳集团www0638院友

个人简介:
袁细国,太阳集团www0638副教授,博士生导师,计算生物信息学研究所副所长,西安市计算生物信息学重点实验室副主任;于2011年6月获西安电子科技大学工学博士学位,并于2009.10-2010.10,2013.01-2013.04在美国弗吉尼亚理工大学做访问学者;从事计算生物信息学、大数据与机器学习、癌症基因组及病原微生物数据分析等研究。主持了国家自然科学基金面上项目、青年基金项目、省自然科学基金项目、企业项目等20余项,在国内外有影响力刊物IEEE TBME,IEEE/ACM TCBB,IEEE TNB,JCB, BMC Genomics等发表学术论文50余篇。



